
Ayuda
InverPep posee actualmente 702 AMPs experimentalmente validados provenientes de diferentes especies de invertebrados de los filos Artrópodos, Moluscos, Nematodos, Anélidos, Equinodermos, Platelmintos, Placozoa, Hydradae y el subfilo Tunicata. Todos estos péptidos se encuentran alojados en la base de datos junto con su información general (filo y especie de origen, nombre, tamaño), las propiedades fisicoquímicas (carga neta, hidrofobicidad, índice de Boman, índice alifático, pI, porcentaje de aminoácidos hidrofóbicos y estructura secundaria, si es conocida), su actividad antimicrobiana y enlaces externos a la bibliografía.
Es una herramienta para calcular diferentes propiedades fisicoquímicas de múltiples péptidos al tiempo, se diseñó usando el
lenguaje de programación PERL (versión 5.22.0.1) y luego se trasladó a PHP para integrarlo a la interfaz web de InverPep.
El programa calcula peso molecular, carga neta a pH neutro, hidrofobicidad, índice de Boman, índice alifático, punto isoeléctrico
y porcentaje de aminoácidos hidrofóbicos.
CALCAMPI requiere que se copien los péptidos listados en la plataforma o se tenga un archivo FASTA con la lista de los péptidos
a ser analizados.
- Peso Molecular: El peso molecular del péptido es la suma de las masas molares de todos los aminoácidos que lo conforman.
- Carga Neta: Para calcular la carga neta se usó la siguiente ecuación:
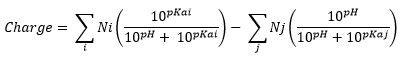
[Nelson, D., & Cox, M. (2008). Lehninger Principles of Biochemistry (5th edition ed.): W. H. Freeman.]
- Hidrofobicidad: La hidrofobicidad se representó en términos del gran promedio del valor de hidrofobicidad, calculado como la suma de los valores hidropáticos de todos los aminoácidos (tomados de la escala Kyte-Doolittle) dividido por el número de residuos de la secuencia. Valores positivos indican péptidos hidrofóbicos y negativos péptidos hidrofílicos.
El porcentaje de aminoácidos hidrofóbicos se calculó en base a la escala Kyte-Doolittle. Se define como la suma de residuos hidrofóbicos del péptido dividido por el número total de residuos.
[Kyte, J., & Doolittle, R. F. (1982). A simple method for displaying the hydropathic character of a protein. Journal of Molecular Biology, 157(1), 105-132.]
- Índice de Boman: El índice de Boman se define como la suma de las energías libres de las cadenas laterales de los aminoácidos al transferirse de ciclohexano a agua dividido por el número de residuos del péptido. Puede servir como criterio para determinar el potencial de un péptido a unirse a una proteína o a una membrana.
[Radzicka, A., & Wolfenden, R. (1988). Comparing the polarities of the amino acids: side-chain distribution coefficients between the vapor phase, cyclohexane, 1-octanol, and neutral aqueous solution. Biochemistry, 27(5), 1664-1670.]
- Índice alifático: El índice alifático es el volumen relativo ocupado por las cadenas laterales alifáticas (alanina, valina, isoleucina y leucina). Valores positivos están asociados a un aumento de la termoestabilidad del péptido. Se calcula con la siguiente ecuación:
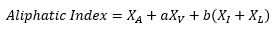
[Ikai, A. (1980). Thermostability and Aliphatic Index of Globular Proteins. Journal of Biochemistry, 88(6), 1895-1898.]
- Punto isoeléctrico: El punto isoeléctrico (pI) es el pH donde la carga neta es igual a cero. El pI se determinó usando un algoritmo en serie que calcula todas las posibles cargas netas al modificar el pH, el programa se detiene cuando encuentra el valor de pH con la carga neta más cercana a 0.
Blast
Blast busca regiones similares entre secuencias. El programa compara secuencias de nucleótidos o proteínas con secuencias de bases de datos y calcula la significancia estadística entre ellos.
http://www.rcsb.org/
[Altschul, S.F., Gish, W., Miller, W., Myers, E.W. & Lipman, D.J. (1990). Basic local alignment search tool. J. Mol. Biol. 215:403-410]
Protein Data Bank (PDB)
PDB es una base de datos de estructuras tridimensionales de moléculas biológicas, tales como proteínas y nucleótidos.
http://blast.ncbi.nlm.nih.gov/Blast.cgi
[H.M. Berman, J. Westbrook, Z. Feng, G. Gilliland, T.N. Bhat, H. Weissig, I.N. Shindyalov, P.E. Bourne (2000). The Protein Data Bank. Nucleic Acids Research, 28: 235-242.]
UniProt
UniProt es un espacio que proporciona información libre, de alta calidad y fácil comprensión de secuencias de proteína y sus funciones.
http://www.uniprot.org/
[The UniProt, C. (2015). UniProt: a hub for protein information. Nucleic Acids Research, 43(D1), D204-D212.]
Expasy
ExPASy es un portal que proporciona bases de datos científicas y herramientas computacionales de diferentes áreas de las ciencias biológicas, incluyendo proteómica, genómica, filogenia, genética poblacional, transcriptómica, etc.
http://www.expasy.org/
[Artimo P, Jonnalagedda M, Arnold K, Baratin D, Csardi G, de Castro E, Duvaud S, Flegel V, Fortier A, Gasteiger E, Grosdidier A, Hernandez C, Ioannidis V, Kuznetsov D, Liechti R, Moretti S, Mostaguir K, Redaschi N, Rossier G, Xenarios I, and Stockinger H (2012). ExPASy: SIB bioinformatics resource portal, Nucleic Acids Res, 40(W1):W597-W603.]
HeliQuest
HeliQuest calcula desde la secuencia de aminoácidos de una hélice (α-hélix, 3-10 hélix, 3-11 hélix o π hélix) sus propiedades fisicoquímicas. Con estos resultados realiza una búsqueda en bases de datos para identificar proteínas que posean segmentos similares.
http://heliquest.ipmc.cnrs.fr/
[Gautier R., Douguet D., Antonny B. and Drin G (2008). HELIQUEST: a web server to screen sequences with specific α-helical properties. Bioinformatics, 24(18):2101-2.]
APD
The Antimicrobial Peptide Database.
http://aps.unmc.edu/AP/main.php
[Wang, G., Li, X. and Wang, Z. (2009) APD2: the updated antimicrobial peptide database and its application in peptide design. Nucleic Acids Research 37, D933-D937.
CAMP
Collection of Anti-Microbial Peptides.
http://www.camp.bicnirrh.res.in/index.php
[Waghu FH, Barai RS, Gurung P, Idicula-Thomas S (2015). CAMPR3: a database on sequences, structures and signatures of antimicrobial peptides. Nucleic Acids Re, gkv1051v1-gkv1051.]
YADAMP
Yet another db of antimicrobial peptides.
http://yadamp.unisa.it/default.aspx
[Piotto, S. P., Sessa, L., Concilio, S., & Iannelli, P. (2012). YADAMP: yet another database of antimicrobial peptides. International Journal of Antimicrobial Agents, 39(4), 346-351.]
- AMP: Antimicrobial Peptides. Péptidos con actividad sobre algún microorganismo. E.g. Cecropinas.
- Actividad: Es la acción que tiene el péptido sobre un grupo de microorganismos. La actividad antimicrobiana puede ser
específica para un solo tipo de microorganismos o puede afectar a más de un grupo de organismos. E.g. Antiviral.
- Código InverPep: código asignado por InverPep para cada uno de sus registros. E.g. 1.
- Especie de origen: especie de donde se obtuvo el péptido. E.g. Drosophila melanogaster
- Estructura secundaria: Plegamiento del péptido debido a las interacciones formadas por las cadenas laterales de los
aminoácidos. E.g. Hélice alfa.
- Filo de origen: Filo al que pertenece la especie de donde se obtuvo el péptido. E.g. Artrópodo.
- Invertebrados: todos aquellos organismos que no se encuentran dentro del subfilo de los vertebrados; es decir, todos
aquellos que carecen de columna vertebral y de esqueleto interno articulado. E.g. Moluscos.
- Nombre del péptido: nombre con el que se encuentra el péptido identificado en la bibliografía. E.g. Big Defensin.
- Secuencia: estructura primaria del péptido. Código de letras asociadas a los aminoácidos. E.g. ESTEVAN.
- Tamaño: Número de aminoácidos que conforman un péptido. E.g. 15 aminoácidos.
Puedes citar InverPep usando la siguiente citación:
Gómez EA, Giraldo P, Orduz S. InverPep: a database of invertebrates antimicrobial peptides. J Glob Antimicrob Resist 2017; 8:13-17.


